武汉康测科技有限公司
9 年
手机商铺
商家活跃:
产品热度:
- NaN
- 0.5
- 0.5
- 1.5
- 0.5
公司新闻/正文
康测金牌产品ChIP-seq,助力老师发文“New Phytologist”
887 人阅读发布时间:2023-10-17 17:02

磷(P)是植物生长和代谢所必需的,是核苷酸、ATP和磷脂等大分子的主要成分。植物主要通过摄取无机磷酸盐(Pi)来获取磷。低磷是限制植物生长和产量的常见非生物胁迫之一。为了应对低磷环境,植物进化出一系列生理生化调节机制,通过调控磷酸盐饥饿响应(PSR)基因的转录激活或抑制而发挥功能。
研究表明,PSR基因的表达受到多种转录因子调控。作者关注的HMGB蛋白具有DNA结合能力,并且是染色质中丰度较高、分布较广的结构蛋白。但水稻中HMGB蛋白的功能和作用机制未知。本研究中,作者借助ChIP-seq、ATAC-seq、RNA-seq等多种组学技术探究了水稻OsHMGB1调控PSR基因表达的作用机制。康测金牌产品ChIP-seq为老师提供助力。
Highlight
丰富的多组学联合结果展示、扎实的组学分析加验证实验
Results
高磷和低磷下OsHMGB1的表达情况
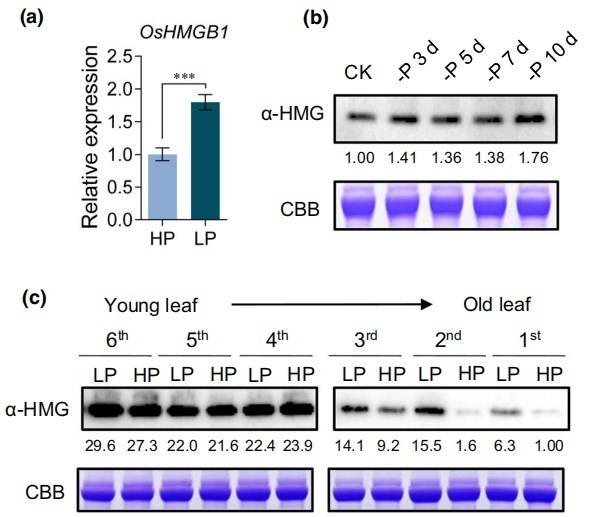
老师们常常会采用转录组测序的方式寻找关键表型与靶标基因表达水平之间的联系。而本研究中,作者目标十分明确(锁定OsHMGB1),首先利用qPCR检测了野生株水稻(WT)在高浓度磷酸盐(HP)和低浓度磷酸盐(LP)条件下OsHMGB1(LOC_Os06g51220)的转录水平(Fig. 1a)。接着,作者使用OsHMGB1抗体(α-HMG)进行WB实验,结果表明:随着磷酸盐(Pi)饥饿时间的延长,OsHMGB1的蛋白水平逐渐升高(Fig. 1b)。并且,与嫩叶相比,老叶中OsHMGB1的蛋白水平在HP和LP条件下差异更明显(Fig. 1c)。综上所述,作者发现Pi受限时OsHMGB1的转录水平和蛋白水平升高。
1、构建转基因植株探究OsHMGB1功能
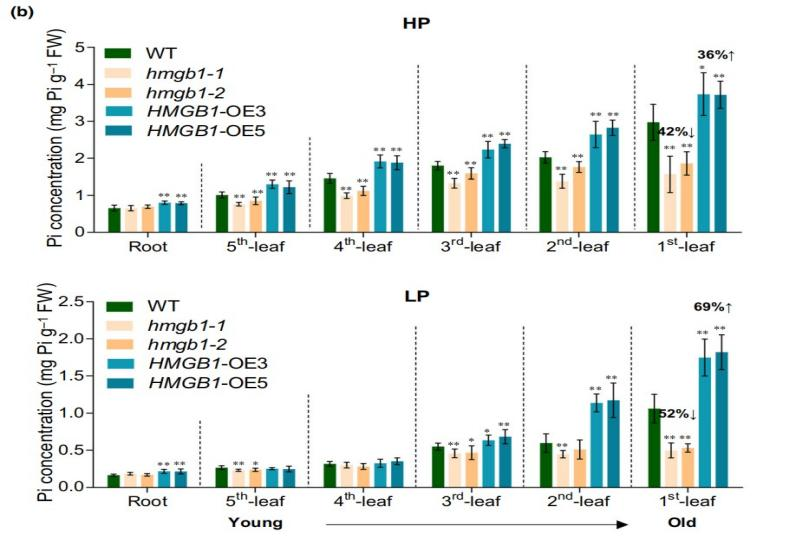
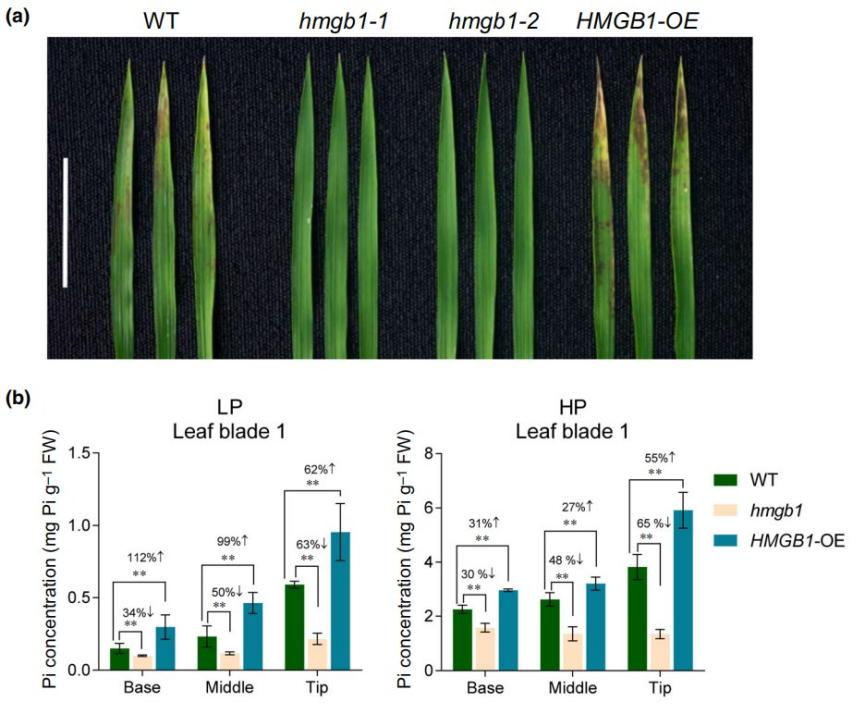
为了进一步研究水稻中OsHMGB1的功能,作者构建了2个OsHMGB1敲除突变株(hmgb1-1、hmgb1-2)和2个OsHMGB1过表达株(HMGB1-OE3、HMGB1-OE5)。温室中,突变株幼苗比WT的生长稍弱,而过表达株幼苗比WT的明显偏小 (Fig. 2a)。大田里,突变株植株比WT的稍短,产量更低。而过表达株则在植株结构和产量上与WT无显著差异。此外,作者发现突变株几乎所有叶子中的Pi浓度均显著低于WT。并且,随着叶龄增加,突变株与WT叶子中的Pi浓度差异越大。相反,过表达株叶子和根中的Pi浓度则明显高于WT(Fig. 2b)。过表达株的老叶尖端出现严重坏死,这是Pi过量的典型症状(Fig. 3a)。
随后,作者分别检测了HP和LP条件下各植株叶子尖端、中部和基部的Pi浓度。结果显示:WT中Pi浓度从基部到尖端逐渐增加。突变株中对应部位的Pi浓度均低于WT,但同一叶子中三个部位的Pi浓度相似。过表达株中对应部位的Pi浓度均明显高于WT,但同一叶子中尖端和基部的Pi浓度差异较大(Fig. 3b)。回补实验证实敲除株的生长缺陷和较低的Pi浓度是由OsHMGB1的功能丧失导致。综上所述,OsHMGB1在水稻Pi稳态和植株生长中发挥重要作用。

2.OsHMGB1蛋白定位在细胞核中
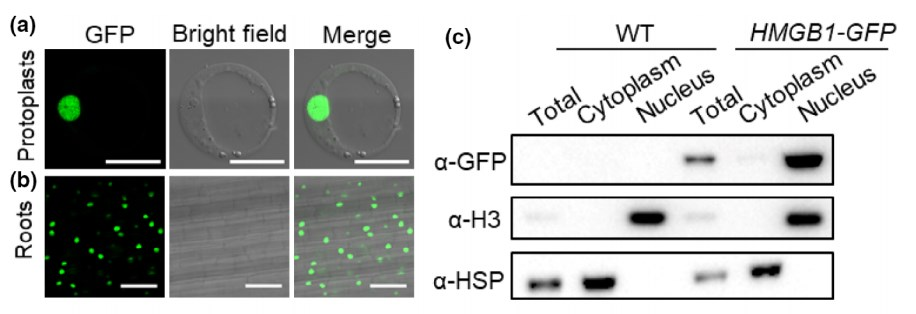
为了确定OsHMGB1的亚细胞定位,作者首先在水稻原生质体中瞬时表达OsHMGB1-GFP融合蛋白(HMGB1-GFP),通过共聚焦激光扫描显微镜在细胞核中检测到GFP荧光信号(Fig. 4a)。并且,在HMGB1-GFP植株的根细胞核中也能观察到GFP荧光信号(Fig. 4b)。随后,作者通过WB实验进一步验证了OsHMGB1的核定位,结果显示OsHMGB1主要在蛋白提取物的细胞核部分,细胞质部分没有(Fig. 4c)。综上所述,OsHMGB1蛋白定位在细胞核中。
3.RNA-seq筛选受OsHMGB1调控的PSR基因
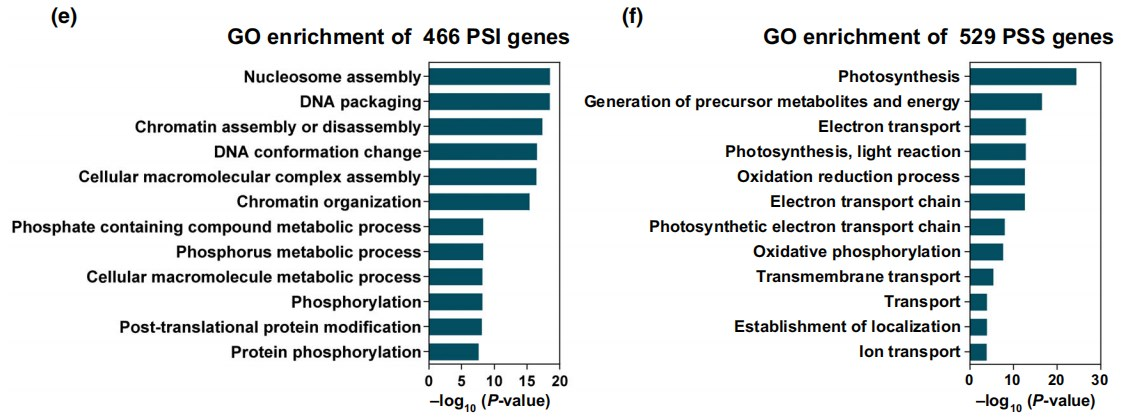
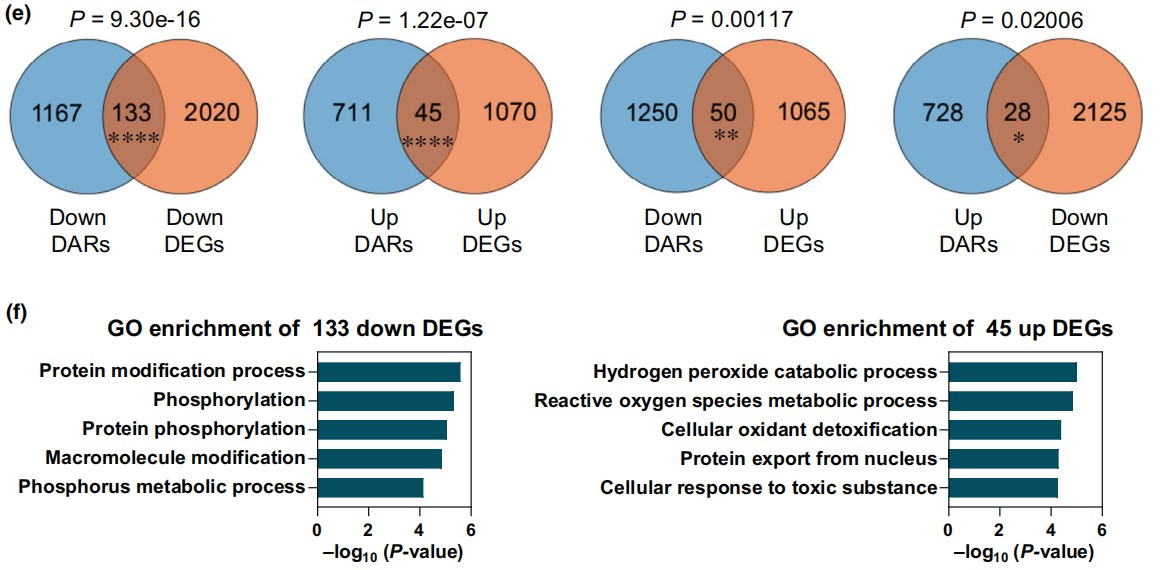
为了探究OsHMGB1调控水稻Pi稳态的机制,作者对HP和LP条件下的WT和hmgb1突变株进行了RNA-seq,分别鉴定到1912和3268个差异表达基因(DEG)(Fig. 5a)。GO分析表明,HP条件下hmgb1突变株中下调表达基因主要在“对刺激的响应”、“蛋白磷酸化”、“对胁迫的响应”和“磷代谢过程”富集(Figs 5b)。

4.ChIP-seq鉴定OsHMGB1结合靶基因
HMGB蛋白具有DNA结合能力,而ChIP-seq是研究蛋白-DNA互作的有利工具,康测ChIP-seq属于金牌产品,提供多重质检报告和个性化分析服务。作者对HMGB1-GFP转基因植株进行了ChIP-seq。作者在三个重复中共鉴定到24907个靶基因,78%的结合位点分布在promoter-TSS区域(Fig. 6a),并且peak在TSS附近显著富集(Fig. 6b)。此外,作者还发现:OsHMGB1结合信号强弱与全基因组表达水平呈正相关(Fig. 6c),并且表达受OsHMGB1调控的基因上OsHMGB1结合信号更强 (Fig. 6d)。
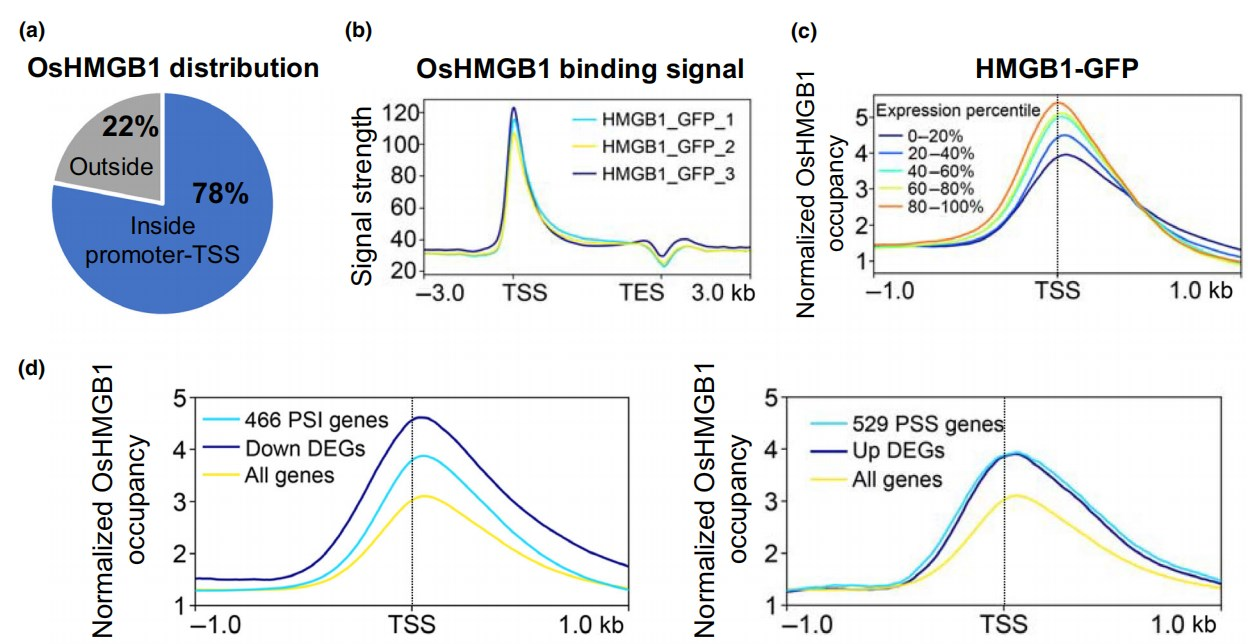
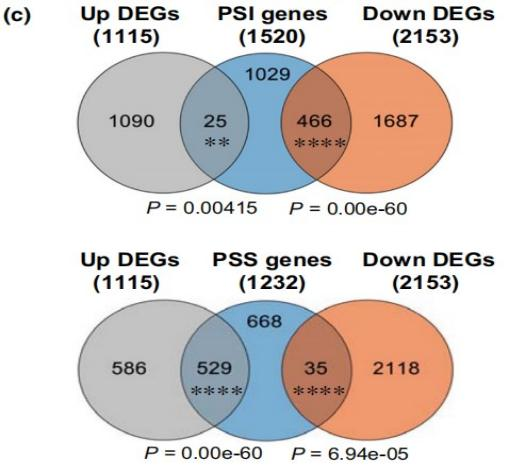
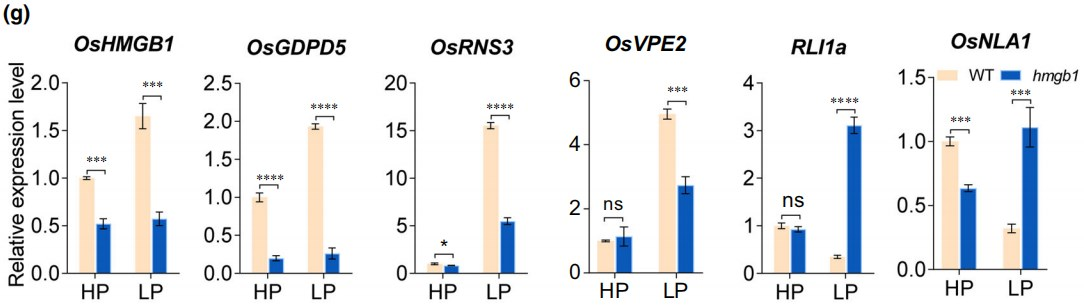
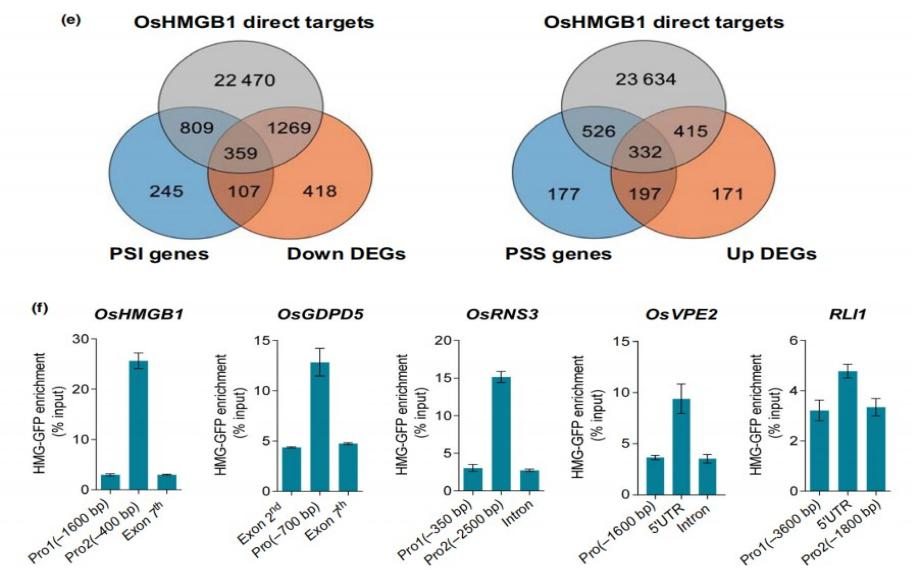
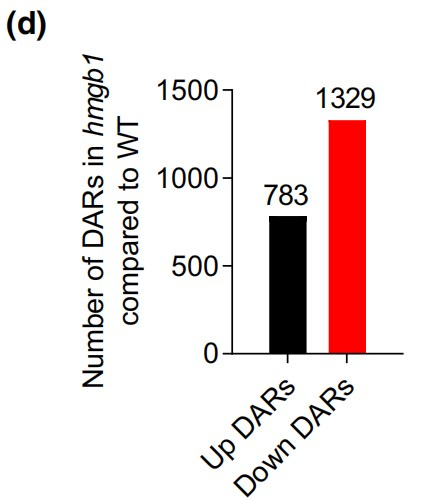
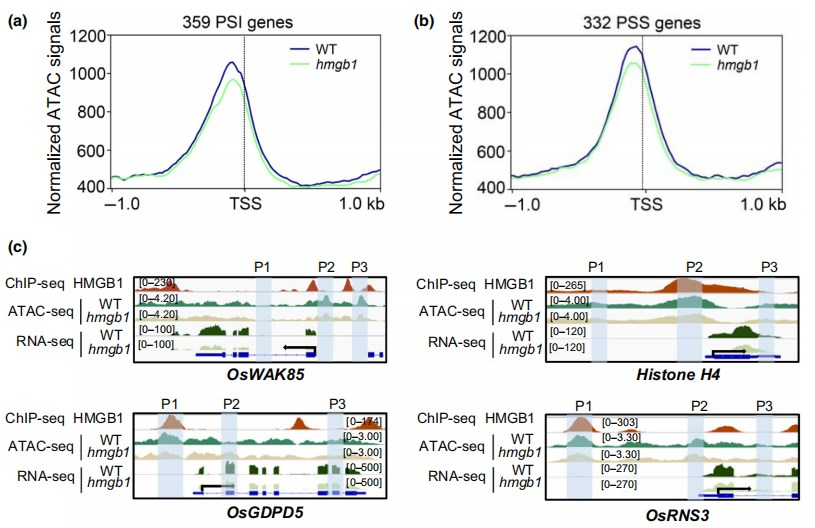
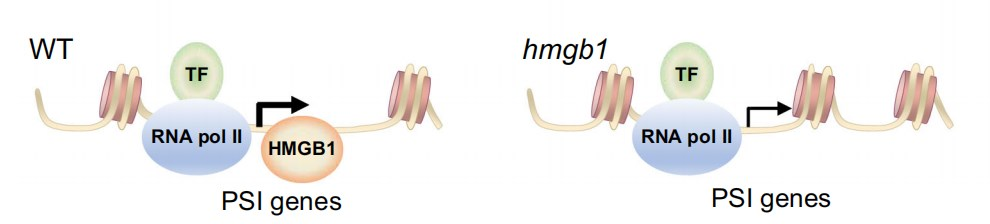
联合ChIP-seq和RNA-seq数据,作者发现有1168个PSI基因也是OsHMGB1直接结合靶基因。其中,有359个PSI基因在hmgb1突变株中表达下调,作者认为这些基因受OsHMGB1直接调控。此外,还有107个PSI基因在hmgb1突变株中表达下调,但不是OsHMGB1直接结合靶基因。因此,这些基因可能受OsHMGB1间接调控(Fig. 6e)。同样地,有332个PSS基因受OsHMGB1直接调控以及197个PSS基因受OsHMGB1间接调控(Fig. 6e)。与ChIP-seq结果一致,ChIP-qPCR证实蛋白OsHMGB1结合基因OsHMGB1、OsGDPD5、OsRNS3、OsVPE2和RLI1的启动子(Fig. 6f)。综上所述,OsHMGB1通过直接结合PSR基因的启动子区来调节基因表达。
5.ATAC-seq检测OsHMGB1对染色质可及性的影响
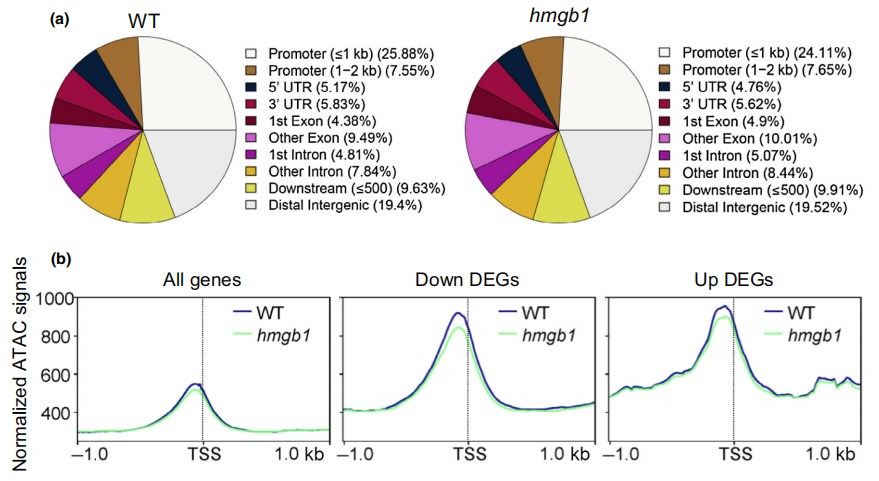
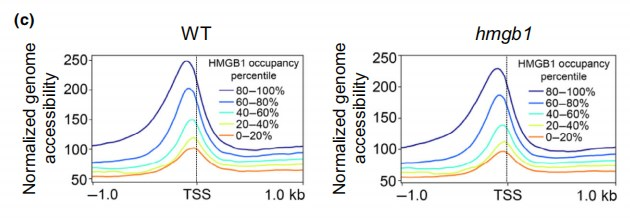
根据注释信息,OsHMGB1是染色质结构蛋白。因此,作者还考虑了OsHMGB1调控过程中可能发生的染色质可及性变化。众所周知,ATAC-seq是研究染色质可及性的利器。因此,作者对WT和hmgb1-1突变株进行了ATAC-seq,并分别在WT和hmgb1突变株中鉴定到44082和47738个peak,其中共有peak 32187个。作者发现peak主要位于启动子区(Fig. 7a),并且hmgb1突变株中基因在TSS附近的染色质可及性信号弱于WT(Fig. 7b),这意味着OsHMGB1的突变降低了染色质可及性。此外,作者还观察到OsHMGB1结合信号强弱与全基因组可及性丰度呈正相关(Fig. 7c)。
通过qPCR和WB,作者发现Pi受限时水稻OsHMGB1的转录水平和蛋白水平升高。
通过构建OsHMGB1敲除突变株、过表达株以及Pi浓度检测,作者发现OsHMGB1在水稻Pi稳态和植株生长中发挥重要作用。
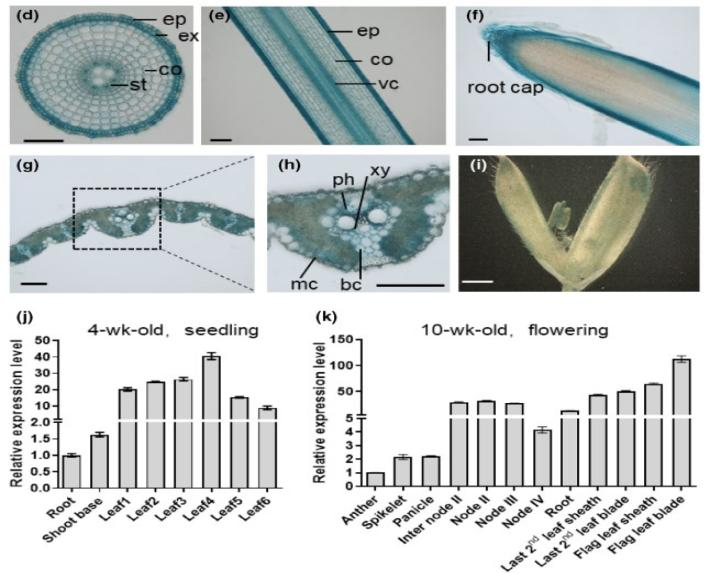
通过检测水稻原生质体中OsHMGB1-GFP融合蛋白的荧光信号,揭示了OsHMGB1的核定位。随后,通过GUS报告基因活性检测和qPCR实验,揭示了OsHMGB1的组织特异性表达。
通过RNA-seq,作者筛选出Pi饥饿响应(PSR)基因,并将其划分为Pi饥饿诱导(PSI)基因和Pi饥饿抑制(PSS)基因两大类。GO分析表明:OsHMGB1正调控的PSI基因主要负责维持染色质结构和Pi稳态,而OsHMGB1负调控的PSS基因主要与光合作用有关。
借助ChIP-seq,作者发现OsHMGB1通过直接结合PSR基因的启动子区来调节基因表达。
通过ChIP-seq、ATAC-seq和RNA-seq联合分析以及MNase-qPCR验证,作者推测OsHMGB1通过减少核小体占位,影响靶标基因启动子区的染色质可及性,调节PSR基因表达,从而调控水稻Pi稳态和植株生长。
参考文献
Yan Wang, Fei Wang, Hong Lu, et al. Rice chromatin protein OsHMGB1 is involved in phosphate homeostasis and plant growth by affecting chromatin accessibility[J]. New Phytologist, 2023